Python отлично подходит для обработки изображений. Сегодня мы расскажем о том, как найти на снимке раковые клетки. На примере реального датасета покажем, как провести фильтрацию (сглаживание) изображения и как найти на нем локальные максимумы с помощью Python-библиотеки skimage.
Датасет изображений
Возьмем датасет (MCF7 Cell Painting) с изображениями клеток, снятых с помощью флуоресцентной микроскопии. Если быть точнее, то изображения представляют собой клетки рака молочной железы MCF-7, обработанные в течение 24 часов набором из 113 небольших молекул в восьми концентрациях.
Скачаем только один архив и распакуем в директорию images/. Если у вас Windows, то качайте напрямую по ссылке, если у вас Unix-подобная ОС, то можете использовать следующие команды (wget можно заменить на curl):
wget https://data.broadinstitute.org/bbbc/BBBC021/BBBC021_v1_images_Week1_22123.zip mkdir images unzip BBBC021_v1_images_Week1_22123.zip -d ./images
Найдем все изображения DAPI
DAPI — это флуоресцентный краситель, который связывается с ДНК и окрашивает ядра на каждом изображении. Обычно сигнал DAPI улавливается в первом канале микроскопа. Поэтому мы будем работать с изображениям с первым каналом. Такие изображения содержать w1 в названии.
Для нахождения файлов по названию используем Python-функцию glob:
from glob import glob
paths = glob('images/Week1_22123/Week1_*_w1*.tif')
paths.sort()
А теперь посмотрим, что за изображения у нас имеются. Пример кода на Python для отображения изображений:
from skimage import io
import matplotlib.pyplot as plt
img = io.imread(paths[0])
print(img.shape, img.dtype, img.min(), img.max())
# => (1024, 1280) uint16 176 10016
plt.figure(figsize=(6, 6))
plt.imshow(img, cmap='gray', vmax=4000)
plt.axis('off')
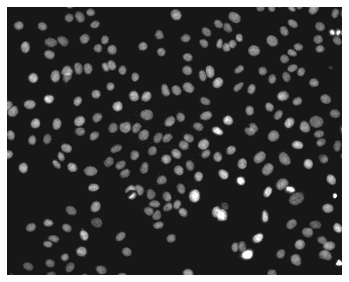
Как найти раковые клетки на изображении с библиотекой skimage
Простой и эффективный способ найти раковые клетки (точнее их центры): сгладить изображение, чтобы уменьшить шум, а затем найти все локальные максимумы, которые выше заданного порога интенсивности. Напишем функцию для этого на нашем примере изображения.
Для работы с изображениями воспользуемся Python-библиотекой skimage [1]. Сначала сгладим изображение с помощью гауссовского фильтра gaussian со значением sigma. Аргумент preserve_range=True не дает провести нормализацию, поэтому сглаженное изображение будет находится в том же диапазон интенсивности, что и входные. Затем найдем локальные максимумы в сглаженном изображении с помощью функции peak_local_max. Установим min_distance=4, чтобы предотвратить нереалистичное сближение любых двух центров, и threshold_abs=1000, чтобы избежать ложных срабатываний в темных областях изображения.
Код на Python для сглаживания и нахождения локальных максимумов выглядит следующим образом:
from skimage.filters import gaussian
from skimage.feature import peak_local_max
def detect_nuclei(img, sigma=4, min_distance=6, threshold_abs=1000):
g = gaussian(img, sigma, preserve_range=True)
return peak_local_max(g, min_distance, threshold_abs)
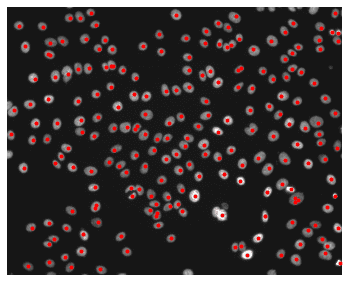
Поссмотрим, как функция определит клетки на изображении выше:
centers = detect_nuclei(img)
plt.figure(figsize=(6, 6))
plt.imshow(img, cmap='gray', vmax=4000)
plt.plot(centers[:, 1], centers[:, 0], 'r.')
plt.axis('off')
В следующей статье эту функцию применим к остальным изображениям с использованием 5 различных методов, включая многопроцессорную обработку, сравним скорость работы и выберем лучший. А о том, как обрабатывать изображения на реальных задачах Data Science вы узнаете на специализированном курсе по компьютерному зрению «VISI: Computer vision на Python» в лицензированном учебном центре обучения и повышения квалификации разработчиков, менеджеров, архитекторов, инженеров, администраторов, Data Scientist’ов и аналитиков Big Data в Москве.



